上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
Amylose 树脂 收藏
Download:
- isoschizomers |
- compatible ends |
- single letter code

上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
ssDNA 接头腺苷酸化的客户,请选用 5´ DNA 腺苷化试剂盒
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
NEBNext® 免疫测序试剂盒(人)
• 通过对受体序列的深入分析,揭示复杂的免疫系统
概述:
免疫组库测序常常用来分析当下的和过去的免疫应答反应。最常应用领域包括:自身免疫性疾病的表征、肿瘤学、传染病中和抗体的发现、肿瘤浸润淋巴细胞以及用作研究残留病的工具。二代测序(NGS)平台在读长和通量方面的最新进展促进了免疫组库测序的发展。
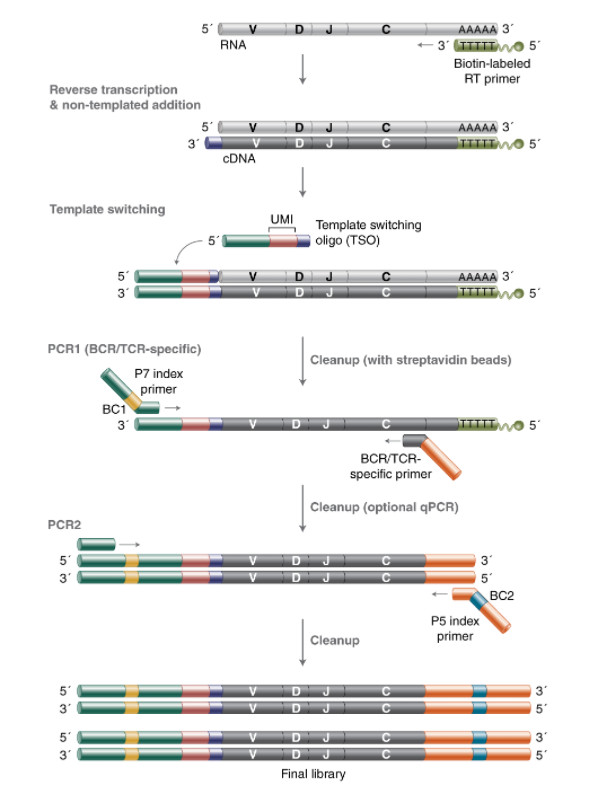
NEBNext® 免疫测序试剂盒(人)通过对 B 细胞和 T 细胞全长免疫组库分析,能获得体细胞的所有突变信息。该试剂盒提供引物模块,可用于分析完整的 V、D、J 区域和 IgM、IgD、IgG、IgA、IgE 亚类,并分析 BCR 轻链、BCR 重链、TCRα 链和 TCRβ 链。该试剂盒还提供分子标签序列(UMI),将来源于同一个分子的 PCR 产物组成共有序列,以便提高测序准确性,消除 PCR 偏差。Galaxy 平台提供一个开源软件工具——pRESTO,用于在本地或云端开展超强生物信息分析。为了确保不熟悉 Galaxy 平台的用户能成功使用分析软件,pRESTO 提供教学教程。
抗体和 TCR 结构的简化示意图:

免疫组建库流程:
产品描述:

使用唯一分子标签序列(UMI)实现克隆型的精准检测。在有或没有 UMI 情况下,比较小鼠 TCR 克隆型频率。根据有 UMI 的测序结果,对前 100 个高表达克隆型进行排序。在有或没有 UMI 情况下,重复建库,克隆型频率重叠绘制于图中。每个条形或圆点代表一个克隆,该克隆经过总读长分析或使用 UMI 过滤。使用 UMI 可对原始样本中的起始 RNA 分子进行绝对定量。当将克隆按照富集度从高到底进行排序,没有 UMI 的数据会导致 PCR 或测序扩增的偏差,从而误导样本中克隆型频率的分析。UMI 的使用通过对起始样本中 RNA 的绝对定量来校正偏差。使用 UMI 也可以在重复本之间实现更好的相关性,尤其是起始量较低时。各个文库向下抽样 500,000 reads。
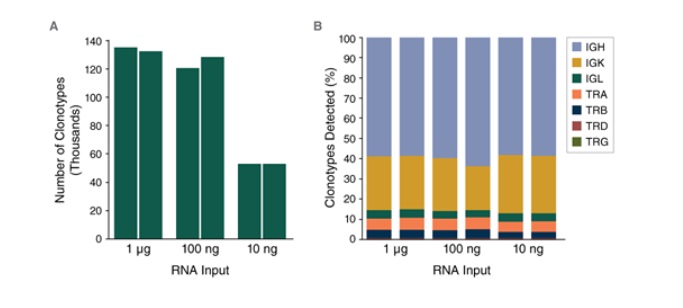
使用 NEBNext 免疫测序试剂盒(小鼠),能够在同一管中制备小鼠 BCR 和 TCR 文库。分别使用 10 ng、100 ng 和 1,000 ng 小鼠脾脏总 RNA(Takara Bio #636605)制备小鼠 BCR+TCR 文库,每个起始量都做有平行实验。所有文库向下抽样 1,000,000 reads。使用 pRESTO 工具完成 reads 过滤、序列组装和基于 UMIs 组成共有序列。使用 MiGMAP 鉴别 V、D 和 J 区域。图(A)表示每个小鼠脾脏总 RNA 起始样本检测到的克隆型数量。图(B)表示各文库中 B 细胞重链(IGH)和轻链(IGK 和 IGL)以及 Tα 链(TRA)、β 链(TRB)、δ 链(TRD)和 γ 链(TRG)所占百分比。
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
The NEBNext® Ultra™ II Directional RNA Second Strand Synthesis Module has been optimized to
generate double-stranded cDNA from first-strand cDNA, as part of the NEBNext Directional RNA library
preparation workflow. This workflow uses the “dUTP” method for strand-specific library construction, which
depends on incorporation of uracil into the second strand cDNA, enabled by the NEBNext Second Strand
Synthesis Reaction Buffer with dUTP Mix, included in this module.
The NEBNext Ultra II Directional RNA Second Strand Synthesis Module is Designed for Use with the
Following:
– NEBNext® Ultra™ II RNA First Strand Synthesis Module (NEB #E7771)
– NEBNext® Ultra™ II End Repair/dA-Tailing Module (NEB #E7546)
– NEBNext® Ultra™ II Ligation Module (NEB #E7595)
– NEBNext® Ultra™ II Q5® Master Mix (NEB #M0544)
– NEBNext® Oligos for Illumina®
#E7535, #E7350)
NEBNext Second Strand Synthesis Enzyme Mix
NEBNext Second Strand Synthesis Reaction Buffer with dUTP Mix
-20°C
请联系我们。 NEBNextSelector.neb.com 可以使用在线产品选择工具进行产品选择。
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
NEBNext 文库稀释缓冲液
提供更精准可靠的定量结果
适用于不同方法制备的多种插入片段大小及 GC 含量的文库
只需要 4 个标准样品,每个试剂盒可定量的文库更多
提供使用方便的文库稀释缓冲液
NEBNext 文库定量预混液只需要加入引物
无论插入片段大小,所有的文库均可使用同一延伸时间
文库定量结果可以通过 NEB 线上工具 NEBioCalculator.neb.com 轻松计算
试剂盒中包含 ROX,方便需要参照染料的 qPCR 仪器进行标准化
二代测序文库的精确定量对于每次测序得 到 的 数 据 量 及 数 据 的 质 量 至 关 重 要 。 特 别是 Illumina 平台,精确的文库定量对于优化簇密度,从而得到最好的测序结果非常重要。实时定量 PCR(qPCR) 是最精确及有效的文库定量方法,与测量总核苷酸浓度的电泳法和分光光度计法相比,qPCR 有更高的一致性和重复性。基于扩增的定量方法只测量包含两个接头序列的分子,从而提供更为精准的可测序文库分子的浓度。
NEBNext 文库定量试剂盒对二代测序中基于 qPCR 的文库定量提供了显着的提升。NEBNext 文库定量试剂盒优化了针对 Illumina 平台基于 qPCR 定量组分。NEBNext 文库定量试剂盒包含 P5 和 P7 Illumina 接头序列的引物,以及一组高质量预稀释的 DNA 标准品,对 150-1000 bp 之间稀释的 DNA 文库进行可靠的定量。
图 1. 使用 NEBNext 文库定量试剂盒定量文库大小浓度,实现最佳簇密度。



– NEBNext 文库定量预混液
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
该产品已停产,推荐替代产品为 NEBNext® Ultra™ II RNA Library Prep Kit for Illumina®(E7770)
– Low input amounts (as low as 10ng total RNA, purified mRNA or ribosomal-depleted RNA)
– Fast workflow (5 – 5.5 hours), with minimal hands-on time
– Robust, reliable performance
– Ultra high fidelity amplification with minimized GC bias
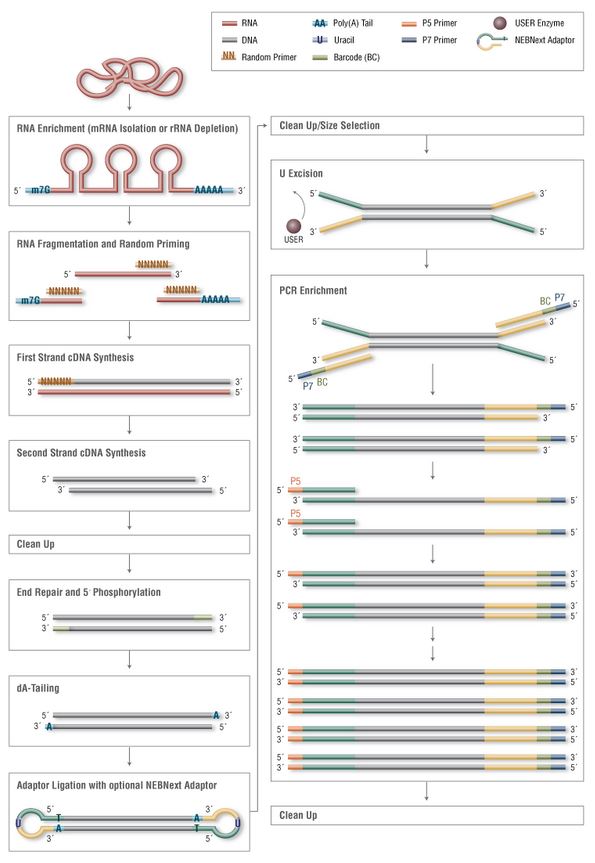
The NEBNext® Ultra™ RNA Library Prep Kit for Illumina® contains reagents for preparation of non-directional RNA libraries for Illumina sequencing. Please note that adaptors, primers, rRNA depletion reagents and poly(A) mRNA isolation reagents are not included in the kit and are available separately.
Ultra Non-directional RNA Library Preparation for Illumina


请联系我们。 NEBNextSelector.neb.com 可以使用在线产品选择工具进行产品选择。
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
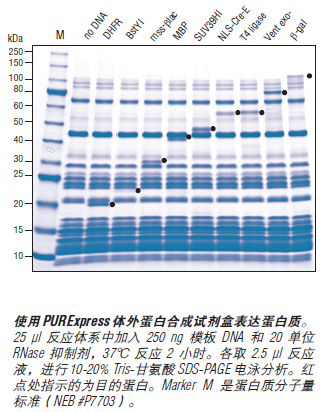
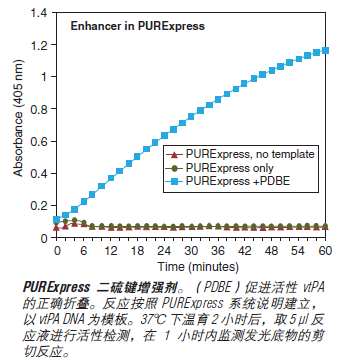
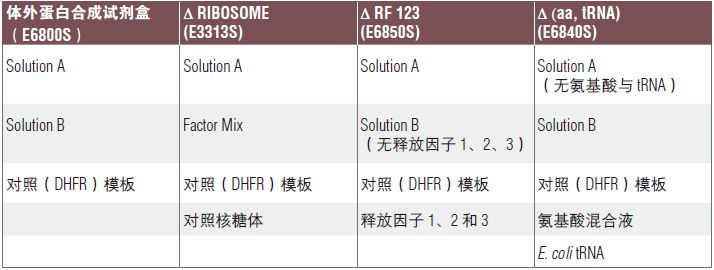
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
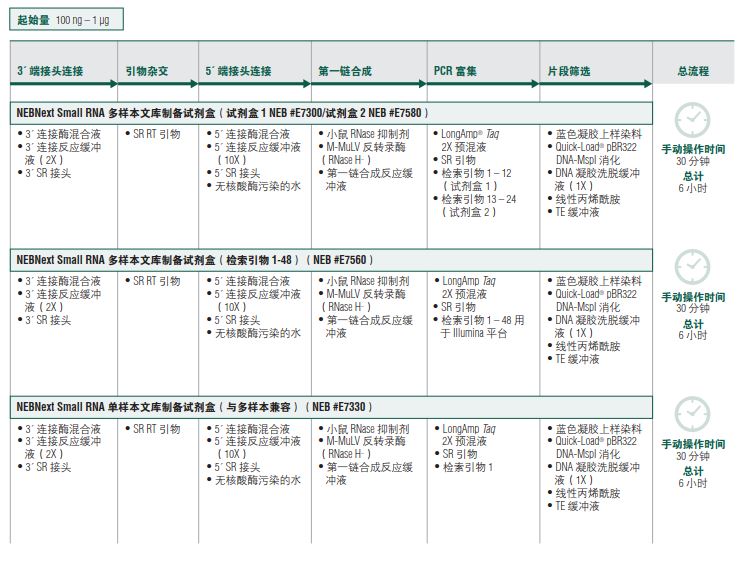
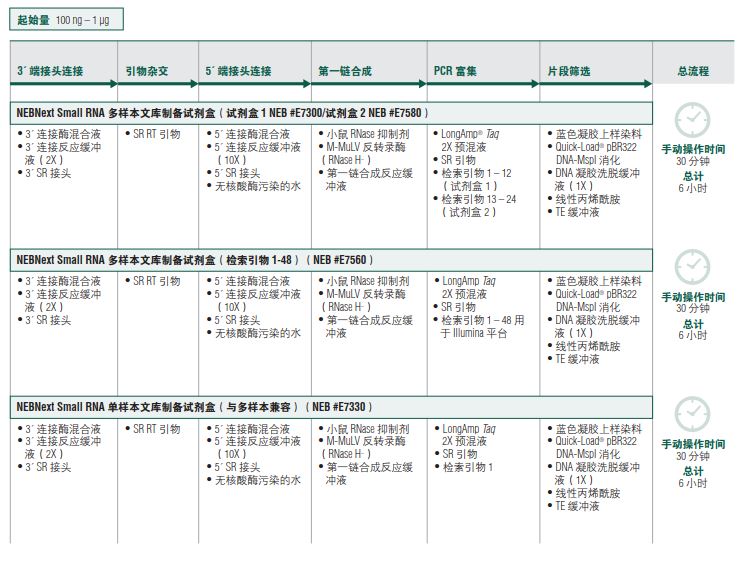
NEBNext Small RNA 多样本文库制备试剂盒 1
NEBNext Small RNA 多样本文库制备试剂盒 2
ssDNA 接头腺苷酸化的客户,请选用 5´ DNA 腺苷化试剂盒
上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
NEBNext 酶学转化法甲基化建库相关产品
NEBNext 酶学转化法甲基化建库试剂盒
NEBNext 甲基化建库酶学转化法模块
NEBNext Q5U™ 预混液
NEBNext 酶学转化法甲基化建库多样本接头引物(双端检索引物)
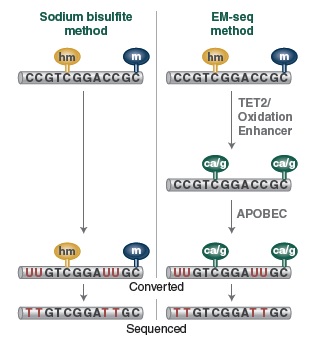
虽然重亚硫酸盐测序是研究 DNA 甲基化的金标准,但这种转化方式会对DNA 造成损伤,导致 DNA 断裂、丢失和 GC 偏嗜。NEBNext 酶学转化法甲基化建库试剂盒 (EM-seq™) 提供了一种酶学方法代替全基因组重亚硫酸盐处理 DNA (WGBS) 的方法,并结合高效流程化的建库步骤,适用于 Illumina 平台测序。高效的 EM-Seq 酶促转化可最大程度地减少对DNA的损伤,结合提供的 NEBNext Ultra II 文库制备流程,最终产生的高质量文库可以从有限的测序数据中更灵敏的检测到 5-mC 和 5-hmC。
更卓越的 5-mC 和 5-hmC 检测灵敏度
更高的比对率
更均一的 GC 覆盖度
能从有限的测序数据检测到更多的 CpG 位点
更均一的二核苷酸分布
更长的文库插入片段
更高效的建库流程
提供转化模块
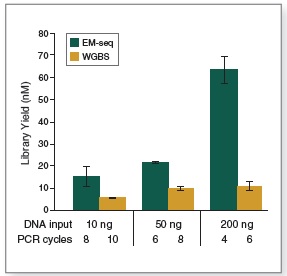
EM-Seq 能得到更高的文库产量。采用Covaris S2 仪器将 10 ng、50 ng 和 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold 试剂盒进行重亚硫酸盐转化。上述所有起始量,EM-Seq 都能使用更少的 PCR 循环得到更高的文库产量,表明 EM-Seq 显著减少了 WGBS 方法中的 DNA 损失。误差条表示标准差。
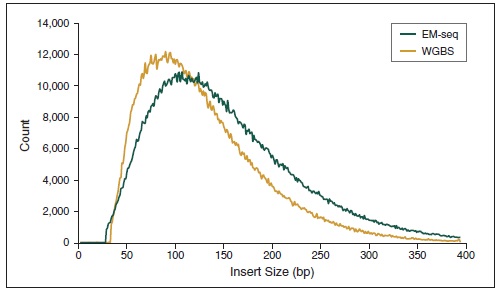
NEBNext 酶学转化法甲基化文库可获得更长的插入片段 。采用Covaris® S2 仪器将 50 ng 人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina MiSeq (2 X 76 bases) 测序,片段大小采用 Picard 2.18.14 测定。图中绘制了每个插入片段出现的频率标准化后的数据,结果如图:EM-Seq 建库后的插入片段比 WGBS 方法得到的插入片段更长,说明:酶学转化法不会对 DNA 造成损伤,而重亚硫酸盐处理的 DNA 有严重损伤。
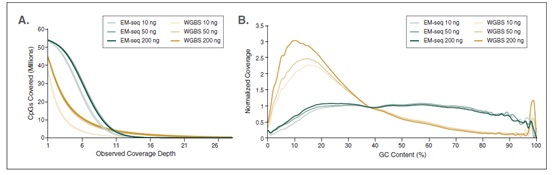
相较于 WGBS,EM-Seq 在更低的测序深度情况下能检测到更多的 CpG 位点,并能实现更卓越的 GC 均一覆盖度。采用Covaris S2 仪器将 10 ng, 50 ng, 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina NovaSeq® 6000 (2 X 100 bases) 测序,使用 bwa-meth 0.2.2 将 测序数据 与 hg38 进行比对。
A: CpG 位点检测:通过分析 3.24 亿双端数据得到 EM-Seq 和 WGBS 文库的 CpG 位点覆盖度,其中每条链都独立计数,最终得到最多 5600 万个可能的 CpG 位点。结果显示:EM-Seq 在更低测序深度能检测到更多的 CpG 位点。
B: GC 覆盖度:使用 Picard 2.17.2 计算 GC 覆盖度,图中显示不同 GC 含量时 (0-100%),标准化后覆盖度的分布情况。结果显示:EM-Seq 文库显著提高 GC 覆盖度的均一性,无 AT 过度测序,也无 GC 测序不足,后两项都是 WGBS 文库的典型缺陷。
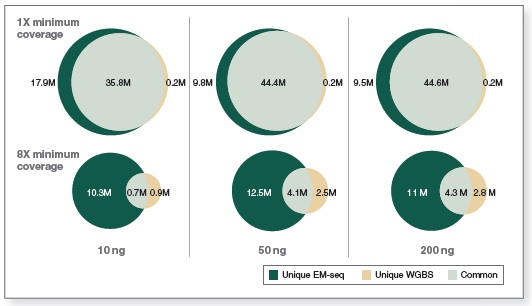
相较于 WGBS,EM-Seq 在更低的测序深度情况下能检测到更多的 CpG 位点。采用 Covaris S2 仪器将 10 ng、50 ng 和 200 ng 不同起始量的人 NA12878 基因组 DNA 打断至 300 bp,同时作为 EM-Seq 和 WGBS 建库的起始样本。对于 WGBS 方法,使用 NEBNext Ultra II DNA 进行建库,随后采用 Zymo Research EZ DNA Methylation-Gold™ 试剂盒进行重亚硫酸盐转化。两个文库均使用 Illumina NovaSeq® 6000(2 X 100 bases)测序,使用 bwa-meth 0.2.2 将测序数据与 hg38 进行比对。通过分析 3.24 亿双端数据得到 EM-Seq 和 WGBS 文库的 CpG 位点覆盖度。
图中显示了 EM-Seq 和 WGBS 两种方法在不同起始量,至少 1X 和 8X 测序深度下检测到的独有和共有的 CpG 位点。EM-Seq 在至少 1X 测序深度下比 WGBS 多检测到 20% 以上的 CpG 位点。而在至少 8X 测序深度下,CpG 位点覆盖度差异增加至 2 倍。