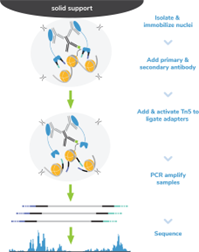
从ChIP-seq到CUT&RUN和CUT&Taq,哪种染色质分析法更适用您的实验?

被广泛使用的传统ChIP-seq与新技术CUT&RUN和CUT&Tag,如何决定哪种染色质分析法更适合您的实验呢?在这里,我们将根据EpiCypher的经验帮您确定最佳检测方法。
关键点1:为何要告别ChIP-seq?
● 样本要上百万个细胞——不适用于珍贵细胞类型或临床样本
● 繁琐的操作步骤——需要交联、染色质片段化和免疫沉淀(IP),实验周期约为一周
● 高测序深度——通常需要每个库2,000 – 4,000万个读段才能在背景上获得足够的信号
● 数据结果不精准 ——背景高,实验重复性差和有非特异性的peak
尽管存在以上短板,ChIP-seq仍然是几十年来应用最广泛的DNA-蛋白互作技术。然而,新方法、新技术往往给科学研究带来天翻地覆的变化,CUTANA™ CUT&RUN和CUT&Tag的出现解决了ChIP-Seq实验需要大量细胞,且重复性差、低信号、高背景等缺点,为研究DNA-蛋白质相互作用提供了新的有效工具。

Figure 1: ChIP-seq与CUT&RUN和CUT&Tag的比较
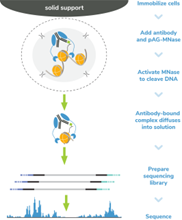
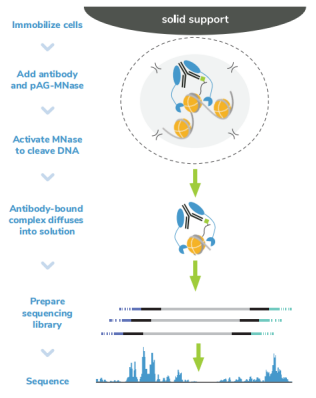
与ChIP-seq相比,CUT&Tag和CUT&RUN具有许多优点。这两种检测方法都不需要交联、染色质片段化或免疫沉淀,即可提供低背景、高可靠性的实验结果。同时CUT&RUN和CUT&Tag的实验周期更短,所需样本细胞更少,测序深度更低。
常见问题
科学方法在不断发展,在表观基因组学领域尤其如此,在过去的十年中,表观基因组学经历了快速的技术增长和扩张。尽管CUTANA™检测具有明显的优势,但许多研究人员对从ChIP-seq转换到CUTANA™仍很犹豫。在这里,我们罗列出可能会在ChIP-seq过渡到CUTANA™ CUT&RUN分析时常见的一些问题。
Q:我正在研究一种瞬态相互作用蛋白质,需要通过交联来稳定染色质上的目标定位。我最好的选择不是ChIP-seq吗?
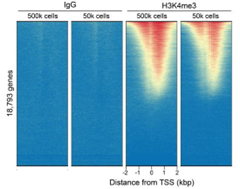
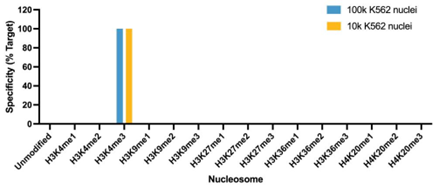
A: CUT&RUN可以生成背景干净的实验数据,免受高度交联相关的可变IP效率的干扰。如果需要,CUTANA检测可与轻到中度交联条件兼容(Fig. 2)。然而,ChIP-seq所需的高度固定方式不能应用于CUT&RUN(或CUT&Tag)。
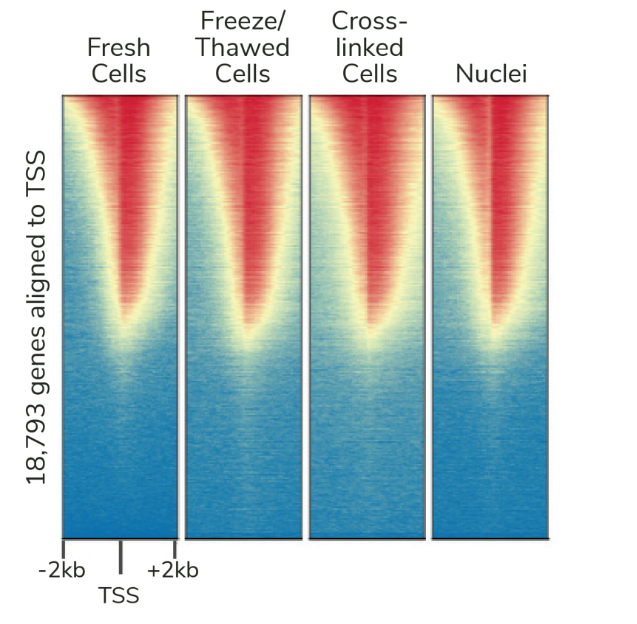
Figure 2: CUT&RUN在样品处理过程中保留了全基因组富集。热图中使用新鲜、冷冻或交联的K562细胞和新鲜细胞核,显示转录起始位点(TSS)的CUT&RUN H3K4me3信号,红色表示H3K4me3高富集。
Q:我正试图将我的结果与已有的ChIP-seq数据进行比较——我需要继续做ChIP-seq吗?
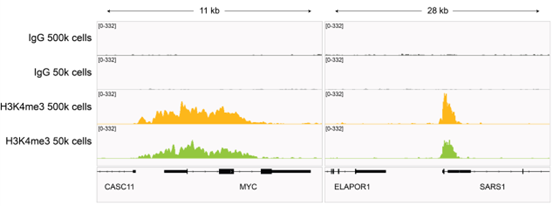
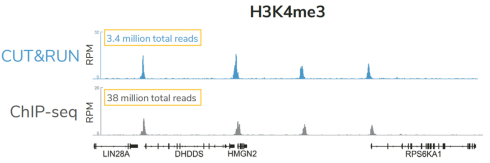
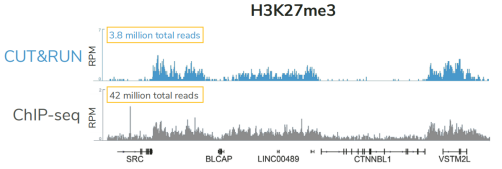
A:虽然ChIP-seq和CUT&RUN是不同的操作步骤,但原始测序数据是相似的,并且使用相同的工具进行处理和可视化。在已有的文献中多次发表过这两种方法的数据比对。主要的区别是CUT&RUN数据的背景要低得多,所需的细胞和测序读段也比ChIP-seq少了10倍。
Q:我已经有了一个很好的ChIP-seq操作方案或有效的抗体,是否应该坚持用下去?
A:与CUT&RUN相比,即使是优化后的ChIP-seq,也需要更多的时间、细胞和测序深度。此外,ChIP-seq本身存在低通量、高背景和成本较高的问题,CUTANA™ CUT&RUN完美的解决了这些问题。与ChIP-seq需要交联、片段化和IP等条件相比,CUT&RUN对大多数目标蛋白和细胞类型的优化需求更低。
Q:由于抗体在ChIP中效果很好,不想换掉ChIP-seq。
A:抗体性能并不是选择ChIP-seq的一个很好的理由。ChIP级别抗体并不可靠,尤其是组蛋白PTMs。EpiCypher发现超过70%的组蛋白赖氨酸甲基化和酰基化PTMs抗体显示明显的交叉反应性和目标蛋白结合效率低的问题。这包括有较高引用率的H3K4me3、H3K9me3、H3K27ac和H3K27me3抗体。非组蛋白PTM靶标,如转录因子,也面临着类似的挑战。
关键点2:CUT&RUN——“万能”染色质分析工具
CUT&RUN是大多数表观基因组实验的理想工具。它为细胞样本、目标蛋白兼容性和测序成本之间提供了很好的平衡。该技术操作非常简单,可根据具体实验情况进行优化调整,且随着EpiCypher开发的CUTANA™ CUT&RUN试剂盒的出现而变得更加容易。
与ChIP-seq相比,CUT&RUN的优点如下:
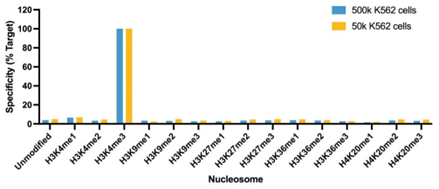
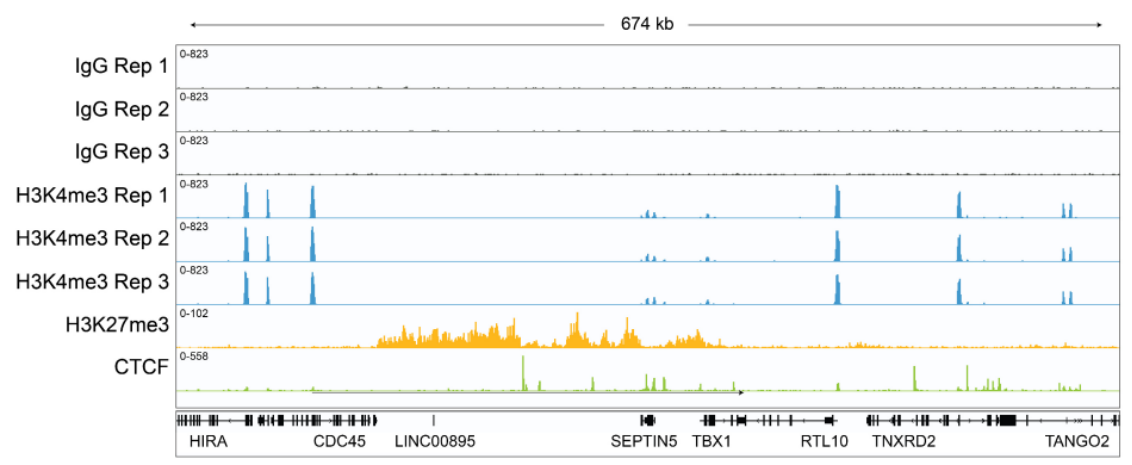
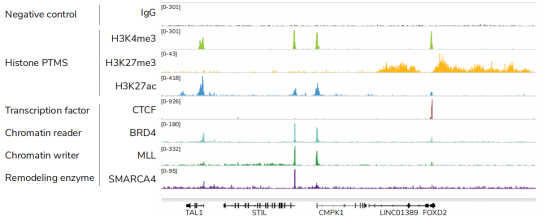
● 针对不同目标蛋白的高分辨率数据:CUT&RUN与组蛋白PTMs和染色质相关蛋白(包括转录因子、 表观遗传学的识别、记录和消除蛋白)兼容,(图3)。 CUT&RUN还可生成很难使用ChIP-seq进行分析的染色质重塑酶图谱,这也突出了CUT&RUN的另一个关键优势。
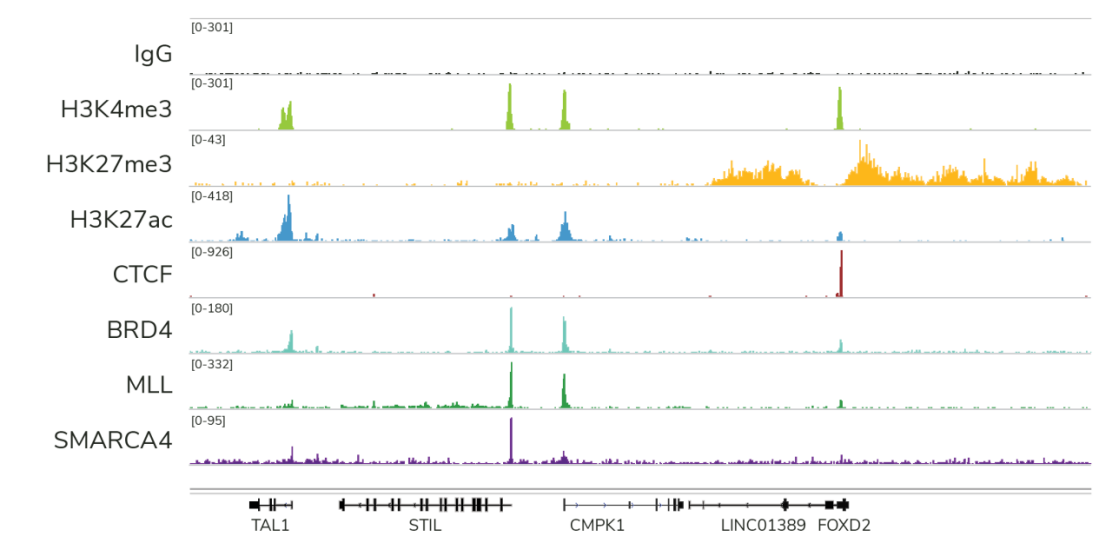
Figure 3: CUTANA™ CUT&RUN分析每次反应仅使用300 – 800万测序读段,为不同的目标蛋白生成高分辨率数据。*每个实验都使用CUTANA™CUT&RUN试剂盒和500,000个K562细胞进行。
● 需要的细胞数量较少:虽然建议使用500,000个以上的细胞,但CUTANA™ CUT&RUN在不改变操作步骤的前提下,可将细胞数量降低至5,000个,从而能够分析不常见的细胞和较珍贵的样本。目前,CUT&RUN已被用于分析小鼠和人类原代细胞、患者来源的异种移植(PDX)、流式细胞仪分选细胞、免疫细胞等。
● 操作步骤简单:CUTANA™ CUT&RUN在3天内即可完成从细胞到文库的建立。还适用于多道移液器和8联排管,提高了分析的重复性和通量。
● 测序成本降低:只需要300 – 800万个测序读段,高通量测序可以检测更多样本。
● 减少实验中需要优化的步骤:如上所述,CUT&RUN跳过了ChIP-seq中最具挑战性的部分(染色质片段化等),只需要较少的优化步骤。EpiCypher的CUTANA™CUT&RUN Kit和CUT&RUN Library Prep Kit使这一过程更加简单。
注:根据EpiCypher的经验,与CUT&Tag相比,CUT&RUN更容易学习和排除故障,特别是在使用EpiCypher的CUT&RUN检测试剂盒和Library Prep试剂盒时。
关键点3:CUT&Tag——“专业级别”染色质分析工具
CUT&Tag更适合在染色质分析测定方面具有经验的研究人员。如果您是:
● 刚刚开始接触表观基因组分析测定
● 经常使用ChIP-seq,打算开始尝试CUTANA染色质分析
● 打算尝试一个新的目标蛋白或使用一个新的细胞类型
● 低丰度目标蛋白,如转录因子和其他染色质相关蛋白
在这些情况中,EpiCypher建议使用CUT&RUN,它有一个简单明了的操作步骤,并可为大多数目标蛋白和细胞类型生成可靠精准的实验结果。
CUT&Tag比CUT&RUN更具挑战性
许多研究人员想要用CUTANA CUT&Tag进行染色质分析实验,因为该方法跳过了传统的文库准备步骤,只需要10万个细胞核即可获得高质量的测序结果。EpiCypher通过的Direct-to-PCR技术进一步简化了CUT&Tag过程,只需要一个管就可完成从细胞到PCR文库扩增。
尽管存在以上优势,根据EpiCypher的经验,CUT&Tag对相关实验操作熟悉度有较高的要求。样品准备不充分或细胞核太少,ConA bead丢失和抗体特异性或效率较低都会影响CUT&Tag的实验结果。与CUT&RUN相比,CUT&Tag也会容易出现更高的duplication rates,并可能在开放染色质区域出现背景信号。基于这些原因,我们推荐大多数用户使用CUT&RUN。
CUT&Tag并不适用于所有目标蛋白
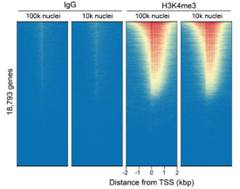
EpiCypher推荐使用CUTANA™ CUT&Tag来研究组蛋白PTMs(图4)和选择转录因子(即CTCF)在全基因组上的结合或分布位点。不建议将CUT&Tag用于染色质相关蛋白分析,这些蛋白通常与染色质结合较弱,在高盐CUT&Tag溶液中剥离。这是该方法的一个主要缺点,也是EpiCypher继续建议大多数用户使用CUT&RUN的原因之一。
注:在ChIP中,样品被交联以稳定染色质上的蛋白质,因此允许使用高盐缓冲液。虽然CUT&Tag与轻度到中度交联兼容,但这些条件严重降低了收率。相反,EpiCypher建议在CUT&RUN中使用新鲜细胞样本。
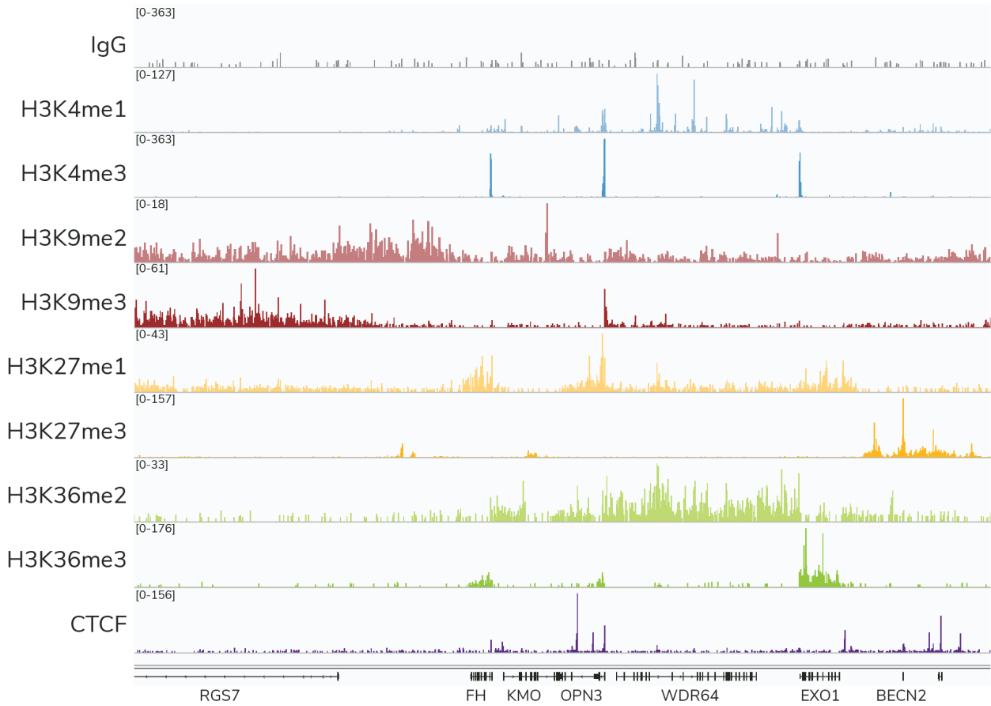
Figure 4: CUTANA™ CUT&Tag是分析组蛋白PTMs的理想工具。
CUT&Tag是低样本量和特殊应用的理想选择
尽管上面列出了一些注意事项,但值得注意的是CUT&Tag是专门为少量细胞的染色质分析而设计的,是CUT&RUN的补充技术。
为什么CUTANA™ CUT&Tag是低样本量应用的理想选择?
● Tn5 tagmentation消除了传统的交联、染色质片段化、IP和文库准备步骤,减少了操作时间并最大化靶标回收率。当尝试用少量或单细胞进行分析时,精简的处理步骤是至关重要的。在CUT&Tag中,pAG-Tn5准确导向结合区域并进行DNA切割,省去了ChIP-seq中最耗时的步骤。
● EpiCypher的Direct-to-PCR CUT&Tag技术允许您在一个管中完成从细胞到PCR文库的扩增。而每次细胞/DNA被洗涤,转移到新的试管中,或在进行纯化时,都会面临丢失样本的风险。Direct-to-PCR CUT&Tag只需要一个DNA纯化步骤,可以在短短两天内完成。
● CUTANA CUT&Tag操作中首选100,000个核,但对于一些选定的目标,可低至1,000个核(图5)。由于CUTANA CUT&RUN分析验证过的最少是5,000个细胞,因此CUT&Tag为研究人员突破表观基因组学的检测界限提供了解决方法。
Figure 5: CUT&Tag仅使用1000个细胞即可生成低丰度(H3K4me3)和高丰度(H3K27me3)组蛋白PTMs的高质量图谱。
选择适合您的染色质分析测定方法
下面是一个快速检查表,可以帮助您为您的项目选择最佳的检测方法:
(一)推荐使用CUT&RUN作为首选的染色质分析检测方法,适用于多种目标蛋白、细胞类型和细胞处理条件。如果每次反应可以有5,000到500,000个细胞,并且满足以下条件,CUT&RUN为最优选择:
1.刚开始接触染色质分析或CUTANA™技术
2.新的目标蛋白或使用新的细胞类型
(二)CUT&Tag是创新型应用于极少量细胞样本的分析方法。适合于有经验的研究人员。
1.CUT&Tag适用于组蛋白PTMs分析
2.CUT&Tag实验条件通常需要比CUT&RUN更多的摸索及优化
3.CUT&Tag每次反应需要1,000至100,000个细胞
References
1. Preissl S et al. Characterizing cis-regulatory elements using single-cell epigenomics. Nat Rev Genet (2022). PubMed PMID: 35840754.
2. Mehrmohamadi M et al. A Comparative Overview of Epigenomic Profiling Methods. Front Cell Dev Biol 9, 714687 (2021). PubMed PMID: 34368164.
3. Carter B et al. The epigenetic basis of cellular heterogeneity. Nat Rev Genet 22, 235-50 (2021 PubMed PMID: 33244170.
4. Agbleke AA et al. Advances in Chromatin and Chromosome Research: Perspectives from Multiple Fields. Mol Cell 79, 881-901 (2020). PubMed PMID: 32768408.
5. Kaya-Okur HS et al. Efficient low-cost chromatin profiling with CUT&Tag. Nat Protoc 15, 3264-83 (2020). PubMed PMID: 32913232.
6. Kaya-Okur HS et al. CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nat Commun 10, 1930 (2019). PubMed PMID: 31036827.
7. Skene PJ et al. Targeted in situ genome-wide profiling with high efficiency for low cell numbers. Nat Protoc 13, 1006-19 (2018). PubMed PMID: 29651053 .
8. Skene PJ et al. An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites. Elife 6, (2017). PubMed PMID: 28079019 .
9. Shah RN et al. Examining the Roles of H3K4 Methylation States with Systematically Characterized Antibodies. Mol Cell 72, 162-77 e7 (2018). PubMed PMID: 30244833.
10. Liu T. Use model-based Analysis of ChIP-Seq (MACS) to analyze short reads generated by sequencing protein-DNA interactions in embryonic stem cells. Methods Mol Biol 1150, 81-95 (2014). PubMed PMID: 24743991.
11. Zang C et al. A clustering approach for identification of enriched domains from histone modification ChIP-Seq data. Bioinformatics 25, 1952-8 (2009). PubMed PMID: 19505939.
12. Evans MK et al. Ybx1 fine-tunes PRC2 activities to control embryonic brain development. Nat Commun 11, 4060 (2020). PubMed PMID: 32792512.
13. Laczik M et al. Iterative Fragmentation Improves the Detection of ChIP-seq Peaks for Inactive Histone Marks. Bioinform Biol Insights 10, 209-24 (2016). PubMed PMID: 27812282.
14. Meers MP et al. Peak calling by Sparse Enrichment Analysis for CUT&RUN chromatin profiling. Epigenetics Chromatin 12, 42 (2019). PubMed PMID: 31300027.
15. Yu F et al. CUT&RUNTools 2.0: A pipeline for single-cell and bulk-level CUT&RUN and CUT&Tag data analysis. Bioinformatics (2021). PubMed PMID: 34244724.
16. Liu N et al. Direct Promoter Repression by BCL11A Controls the Fetal to Adult Hemoglobin Switch. Cell 173, 430-42 e17 (2018). PubMed PMID: 29606353.
17. de Bock CE et al. HOXA9 Cooperates with Activated JAK/STAT Signaling to Drive Leukemia Development. Cancer Discov 8, 616-31 (2018). PubMed PMID: 29496663.
18. Janssens DH et al. Automated in situ chromatin profiling efficiently resolves cell types and gene regulatory programs. Epigenetics Chromatin 11, 74 (2018). PubMed PMID: 30577869.
19. Uyehara CM et al. Direct and widespread role for the nuclear receptor EcR in mediating the response to ecdysone in Drosophila. Proc Natl Acad Sci U S A 116, 9893-902 (2019). PubMed PMID: 31019084.
20. Zhang XL et al. Reorganization of postmitotic neuronal chromatin accessibility for maturation of serotonergic identity. Elife 11, (2022). PubMed PMID: 35471146.
21. Wang J et al. EZH2 noncanonically binds cMyc and p300 through a cryptic transactivation domain to mediate gene activation and promote oncogenesis. Nat Cell Biol 24, 384-99 (2022). PubMed PMID: 35210568.
22. Hainer SJ et al. Profiling of Pluripotency Factors in Single Cells and Early Embryos. Cell 177, 1319-29 e11 (2019). PubMed PMID: 30955888.
23. Mathsyaraja H et al. Max deletion destabilizes MYC protein and abrogates Emicro-Myc lymphomagenesis. Genes Dev 33, 1252-64 (2019). PubMed PMID: 31395740.
24. Roth TL et al. Reprogramming human T cell function and specificity with non-viral genome targeting. Nature 559, 405-9 (2018). PubMed PMID: 29995861.
25. Collins PL et al. DNA double-strand breaks induce H2Ax phosphorylation domains in a contact-dependent manner. Nat Commun 11, 3158 (2020). PubMed PMID: 32572033.
26. Yusufova N et al. Histone H1 loss drives lymphoma by disrupting 3D chromatin architecture. Nature 589, 299-305 (2021). PubMed PMID: 33299181.
27. Janssens DH et al. Automated CUT&Tag profiling of chromatin heterogeneity in mixed-lineage leukemia. Nat Genet 53, 1586-96 (2021). PubMed PMID: 34663924.
28. Henikoff S et al. Efficient chromatin accessibility mapping in situ by nucleosome-tethered tagmentation. Elife 9, (2020). PubMed PMID: 33191916.
29. Deng Y et al. Spatial-CUT&Tag: Spatially resolved chromatin modification profiling at the cellular level. Science 375, 681-6 (2022). PubMed PMID: 35143307.
30. Gopalan S et al. Simultaneous profiling of multiple chromatin proteins in the same cells. Mol Cell 81, 4736-46 e5 (2021). PubMed PMID: 34637755.
31. Xiong H et al. Single-cell joint detection of chromatin occupancy and transcriptome enables higher-dimensional epigenomic reconstructions. Nat Methods 18, 652-60 (2021). PubMed PMID: 33958790 .
32. Zhu C et al. Joint profiling of histone modifications and transcriptome in single cells from mouse brain. Nat Methods 18, 283-92 (2021). PubMed PMID: 33589836.
33. Janssens DH et al. CUT&Tag2for1: a modified method for simultaneous profiling of the accessible and silenced regulome in single cells. Genome Biol 23, 81 (2022). PubMed PMID: 35300717.
34. Wu SJ et al. Single-cell CUT&Tag analysis of chromatin modifications in differentiation and tumor progression. Nat Biotechnol (2021). PubMed PMID: 33846646.
35. Bartosovic M et al. Single-cell CUT&Tag profiles histone modifications and transcription factors in complex tissues. Nat Biotechnol (2021). PubMed PMID: 33846645.
如需了解更多详细信息或相关产品,请联系EpiCypher中国代理商-上海金畔生物