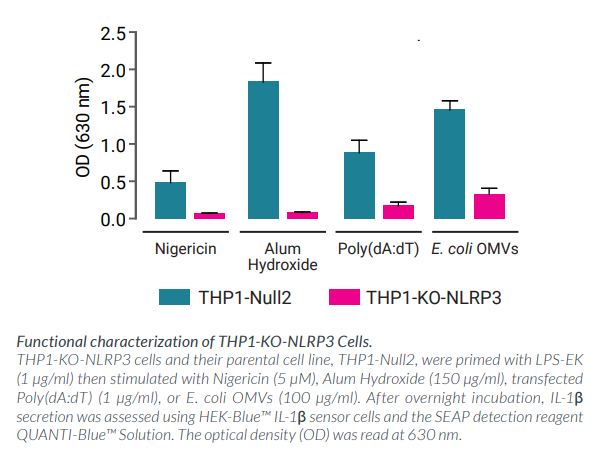
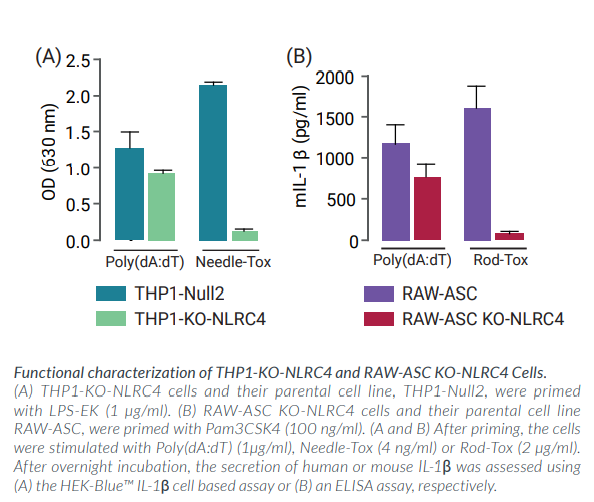
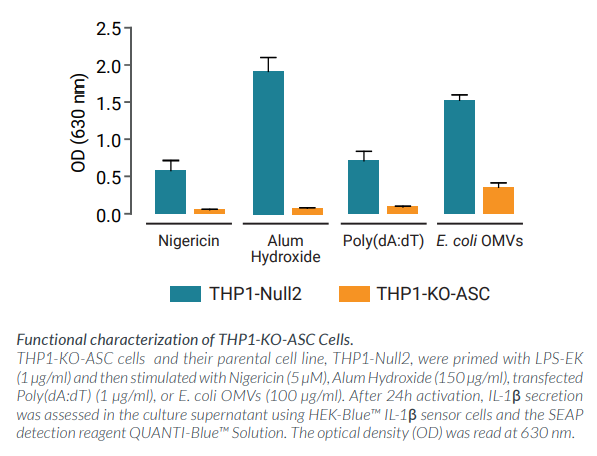
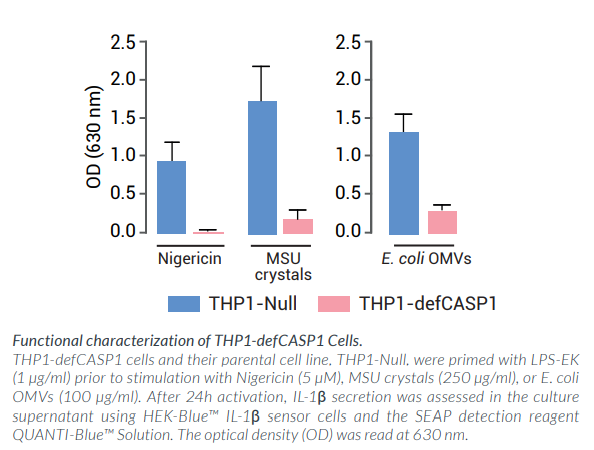
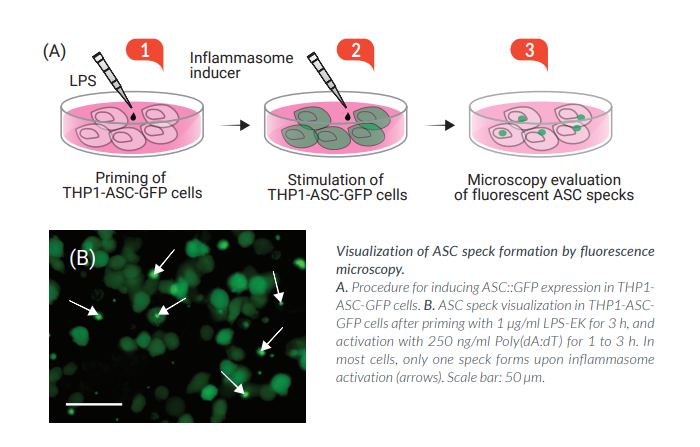
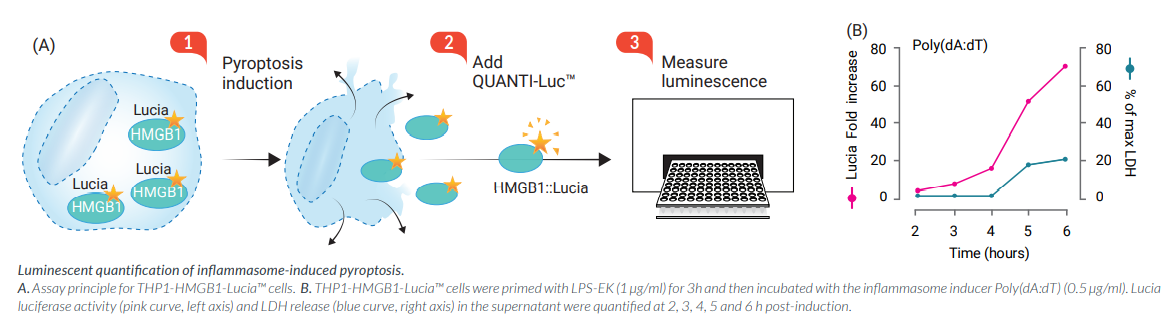
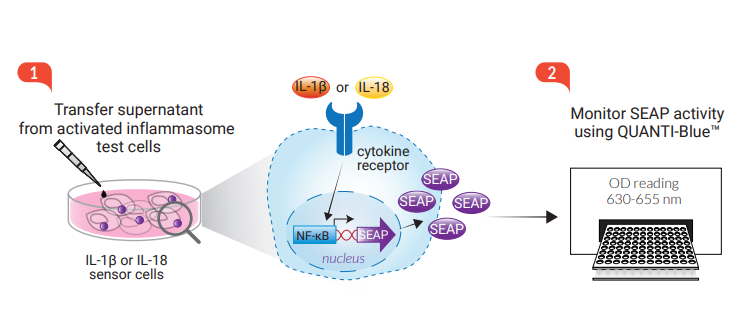
炎症小体的激活与组装
炎症小体依赖于一个“两步”活化模型。不过每个炎症小体都有独特的传感器激活和平台关联机制。这些传感器既可以直接与它的激动剂结合,也可以依赖于另一种分子。ASC接头蛋白对于某些炎症小体的组装是必需的,但也可以不参与或完全缺乏于某些炎症小体的组装。不同炎症小体之间的相互作用通过特定的保守结构域发生。各种激活和组装策略可能经进化,演变成能对威胁做出及时的响应。
“两步”激活模型
启动(Priming)和激活是所有炎症小体都需要的两个步骤。首先,启动步骤可以视为一种必要的调节,以避免不必要的激活。它允许炎症小体组装和下游信号所需蛋白的转录上调。一旦启动,传感器将保持在自动抑制但对信号敏感的状态。它们通过直接的配体结合或间接的细胞内事件被不同的PAMPs或DAMPs激活。
第二步激活,是诱导启动后的传感器进行翻译后修饰(PTMs),导致去抑制的构象变化。这是传感器寡聚化和炎症小体平台组装的起点。下文我们详细介绍了NLRP3、NLRC4和CASP-11/4/5炎症小体的独特激活和组装机制。
NLRP3炎症小体
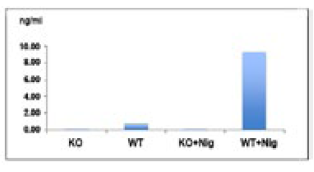
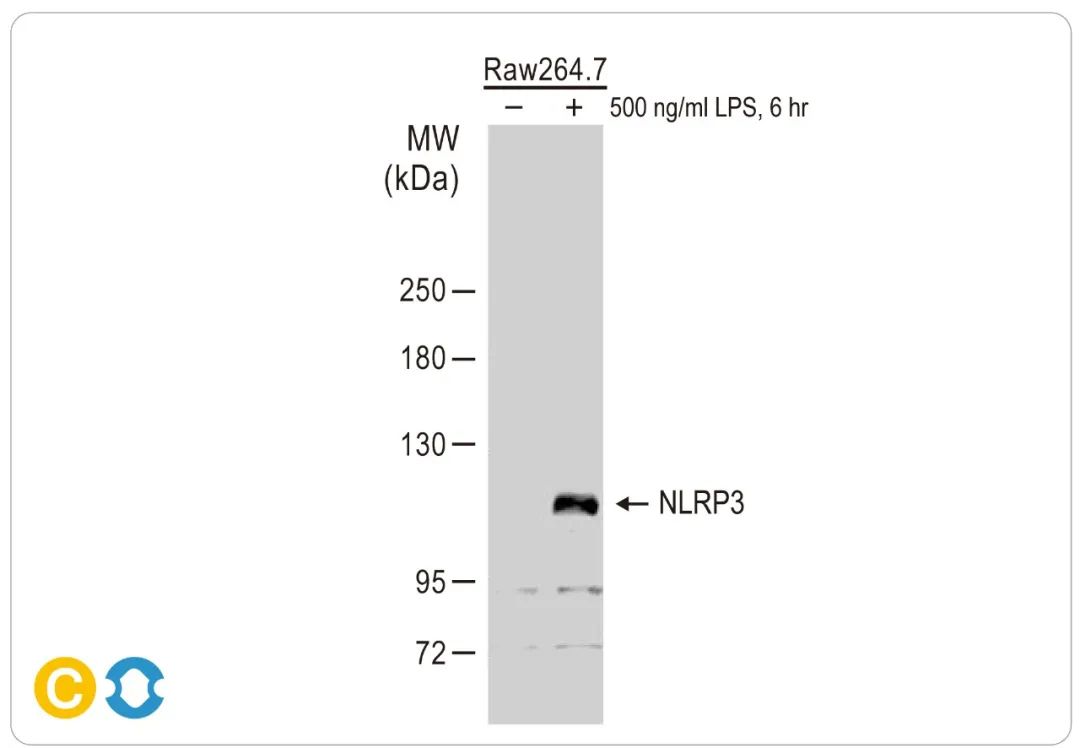
NLRP3炎症小体是最被研究透彻的经典炎症小体。然而,关于NLRP3激活的确切机制仍存在争议 [5, 38]。NLRP3可以被一系列结构上和化学上无关的刺激物激活。这些刺激物可以是无菌的(例如尿酸晶体、β-淀粉样蛋白、胆固醇晶体)、来自微生物(例如造孔毒素、离子通道激活剂)或环境的(例如石棉、二氧化硅)[5]。目前的认知是NLRP3不直接与这些分子结合。相反,它感知下游细胞溶质应激信号,如K+ 外排 [5, 38]、外化线粒体心磷脂和氧化线粒体DNA,后两者在一些研究中被认为是NLRP3的“最终”配体 [39-41]。
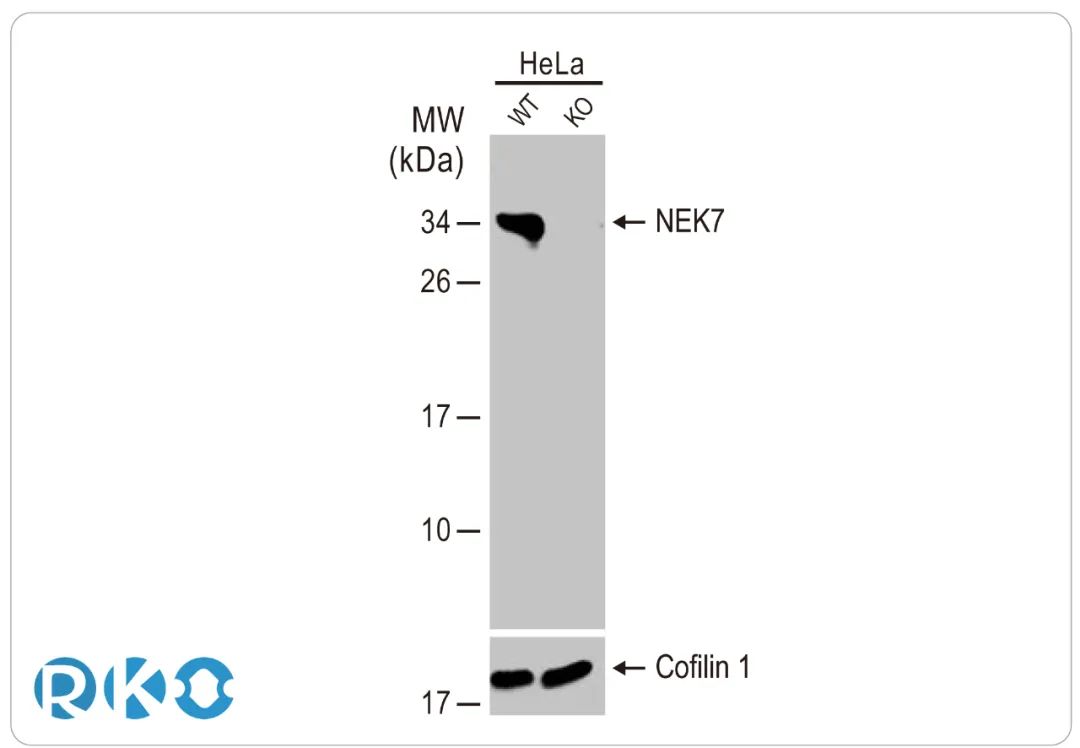
NLRP3是一种拥有三个结构域的蛋白,包含LRR、NOD(或NATCH)和PYD结构域(见第4页)。由于NOD和LRR结构域之间的内部相互作用,NLRP3被自动抑制为闭合构象 [42]。NOD结构域的ATP结合和水解允许NLRP3变成开放构象 [43]。此外,PTMs由不同的刺激物驱动,这些刺激物可以靶向NLRP3结构域中的特定残基 [38, 44, 45]。例如PRR配体(如LPS或Pam3CSK4)的刺激,可以在NOD结构域中导致JNK1介导的S198磷酸化以及NLRP3去泛素化 [44]。一旦LRR结构域被暴露,它就与NEK7(NimA相关蛋白激酶7)结合。NEK7对于桥接相邻NLRP3亚单位之间的间隙是必需的,不过,这种相互作用发生的时间和位置尚未完全阐明 [25-27]。NLRP3的激活会使炎症小体开始组装。NLRP3-PYD结构域招募ASC,然后通过其CARD结构域与pro-caspase-1结合。CASP-1受邻近诱导进行自溶性活化,从而使细胞表面形成GSDMD孔,允许IL-1β/IL-18和警报素分泌,最终导致细胞焦亡 [5, 46]。
细胞器在NLRP3炎症小体组装中起关键作用 [5, 47]。启动和激活信号一同协调NLRP3炎症小体成分在线粒体、内质网(ER)和高尔基体的亚细胞定位。至于是否存在多种细胞器的机制去调控NLRP3炎症小体组装则还在研究中 [48]。
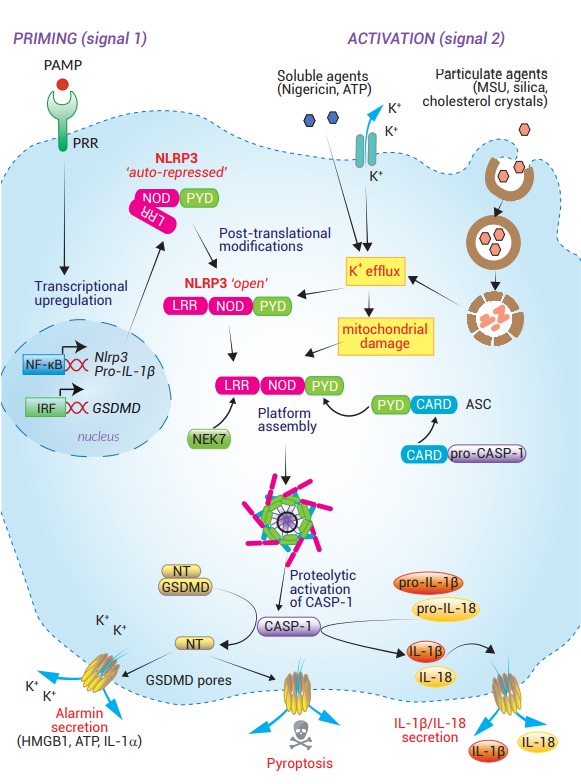
NLRP3炎症小体已被发现有不同的启动机制。长时间的TLR刺激(>3小时)可以触发NF-κB介导的新NLRP3分子转录 [44, 49]。短时间的TLR刺激(<1小时)较可能触发MyD88或TRIF介导的NLRP3蛋白PTM [44, 50]。PTM介导的NLRP3启动可能已经进化到允许即时的炎症反应发生。另外,转录介导的启动可以通过上调NLRP3和效应分子的表达,来增强炎症反应。近期也有研究显示,至少在体外的人类单核细胞中,TLR介导的启动对于NLRP3炎症小体的活化并不是必需的 [51]。
重要的是,非经典炎症小体CASP-11/4/5炎症反应也能触发NLRP3炎症小体的激活和组装(见下文)。
NLRC4/NAIP炎症小体
NLRC4感测细胞内细菌分子,如属于运动器官的鞭毛蛋白,或来自细菌III型或IV型分泌系统(T3SS或T4SS)的内杆和针状蛋白。更具体地说,NAIP与其相应的配体直接结合后,再结合NLRC4。另外,人源和鼠源的NAIP表达存在差异。人源基因组只表达一种NAIP,作用于NLRC4上游并与上述激活剂结合 [7, 52]。这种独特的人源NAIP存在两种异构体,且对感知鞭毛蛋白和T3SS蛋白有不同的亲和力 [52]。而小鼠表达多个NAIPs,它们对每个配体都表现出不同的亲和力;例如NAIP1对T3SS针状蛋白具有较高的亲和力,NAIP2对T3SS内杆蛋白具有较高的亲和力,NAIP5和NAIP6对鞭毛蛋白具有较高的亲和力 [8-11]。尽管NAIPs和NLRC4的转录调控仍缺乏文献记载,但最近发现鼠源NAIPs的本底表达至少依赖于IRF8 [53] 。

NAIPs和NLRC4拥有相似的LRR和NOD结构域,其中LRR结构域是令它们处于自抑制状态所必需的 [8, 54-56]。此外,NLRC4还有一个CARD 结构域。一旦配体结合到NAIP的NOD区域,处于自抑制状态的NAIP就会伸展出来,暴露出NOD结构域 [55]。被激活的NAIP通过NOD-NOD相互作用募集NLRC4启动子(promoter),迫使NLRC4开放。而活化的NLRC4利用其暴露出来的NOD表面与其他NLRC4分子发生骨牌反应,形成组装炎症小体平台 [56]。NLRC4的CARD结构域聚集后会使NLRC4聚合化,NLRC4/NAIP复合物然后可以通过直接的CARD-CARD相互作用或通过ASC接头蛋白与pro-caspase-1结合 [15, 56]。
CASP-11和CASP-4/5非经典炎症小体
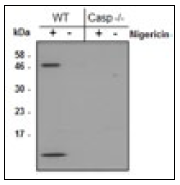
鼠源CASP-11和人源CASP-4/5同时作为传感器和效应蛋白分子,形成非经典炎症小体。CASP-11是在感染革兰氏阴性细菌小鼠模型中首次被发现能感应细胞内的LPS,且不依赖于细胞表面的LPS受体TLR4 [57, 58]。此外,CASP-11和CASP-4/5会驱动GSDMD介导的细胞死亡和CASP-1依赖性的IL-1β/IL-18分泌 [22]。
LPS的识别和caspase的寡聚化都是被CASP-11/4/5的CARD结构域所介导,它们也是使其自身亚单位进行自催化激活所必需的 [29]。这些活化的caspase会切割GSDMD,最终诱导细胞焦亡的发生。与CASP-1不同,CASP-11/4/5不会将pro-IL-1β和pro-IL-18切割成成熟形态 [59]。当质膜形成GSDMD孔,会释放出作为应激信号的胞浆成分。K+外排会促使NLRP3炎症小体组装和CASP-1介导的IL-1β/IL-18分泌 [38, 60]。不过,关于CASP-11/4/5确切的激活机制仍需要进行深入的研究。
细胞外LPS通过细菌OMV的摄取(见第13页)或细胞内细菌的溶解进入胞质 [57, 61]。之后的事件会涉及鸟苷酸结合蛋白(GBPs)和干扰素反应基因B10蛋白(IRGB10)的参与。GBPs积聚在吞噬体膜上,促使细菌产物(如DNA、LPS)释放到细胞质中 [62]。它们会通过招募IRGB10参与穿透细胞内细菌膜 [63]。GBPs也被认为能促使CASP-11与LPS疏水脂质A的相互作用 [64]。
人源CASP-4/5和鼠源CASP-11的本底表达会因细胞类型而有所不同,它们的表达可在使用各种TLR激动剂(如LPS和Pam3CSK4)和干扰素(IFN-β和IFN-γ)预处理后获得上调 [22, 65-67]。同样,GBPs和IRGB10也受干扰素转录调控 [67]。
既然人类基因存在着两种CASP-11相似的蛋白,这就引申出CASP-4和CASP-5在功能上是否具冗余性的问题。目前有体外实验显示要应答转染的LPS,最低限度也需要CASP-5,但在小鼠感染伤寒沙门氏菌的情况下CASP-5则是必需的 [68]。CASP-4和CASP-5在体内细菌感染中的确切作用还待进一步研究。
更多资料或相关产品信息请咨询Invivogen中国代理商-上海金畔生物
参考资料:
5. Swanson, K.V., M. Deng, and J.P.Y. Ting, Nature Reviews Immunology, 2019
7. Yang, J., et al., Proceedings of the National Academy of Sciences, 2013. 110(35): p. 14408-14413.
8. Kofoed, E.M. and R.E. Vance, Nature, 2011. 477(7366): p. 592-595.
9. Zhao, Y., et al., Nature, 2011. 477(7366): p. 596-600.
10. Rauch, I., et al., Journal of Experimental Medicine, 2016. 213(5): p. 657-665.
11. Zhao, Y., et al., Journal of Experimental Medicine, 2016. 213(5): p. 647-656.
15. Xue, Y., et al., Trends in Immunology, 2019. 40(11): p. 1035-1052
22. Kayagaki, N., et al., Nature, 2011. 479(7371): p. 117-121.
25. He, Y., et al., Nature, 2016. 530: p. 354
26. Sharif, H., et al., Nature, 2019. 570(7761): p. 338-343.
27. Thornberry, N.A., et al., Nature, 1992. 356(6372): p. 768-774.
29. Man, S.M. and T.-D. Kanneganti, Nature Reviews Immunology, 2015. 16(1): p. 7-21.
38. Yang, Y., et al., Cell Death & Disease, 2019. 10(2): p. 128.
39. Iyer, S.S., et al., Immunity, 2013. 39(2): p. 311-323.
40. Elliott, E.I., et al., The Journal of Immunology, 2018. 200(9): p. 3047-3052.
41. Zhong, Z., et al., Nature, 2018. 560(7717): p. 198-203.
42. Tschopp, J. and K. Schroder, Nature Reviews Immunology, 2010. 10(3): p. 210-215.
43. Duncan, J.A., et al., Proceedings of the National Academy of Sciences, 2007. 104(19): p. 8041-8046.
44. Groslambert, M. and B.F. Py, J Inflamm Res, 2018. 11: p. 359-374.
45. Kelley, N., et al., International Journal of Molecular Sciences, 2019. 20(13): p. 3328.
46. Platnich, J. and D. Muruve, Archives of Biochemistry and Biophysics, 2019. 670.
47. Hamilton, C. and P. Anand, . F1000Research, 2019. 8(676).
48. Chen, J. and Z.J. Chen, Nature, 2018. 564(7734): p. 71-76.
49. Bauernfeind, F.G., et al., The Journal of Immunology, 2009. 183(2): p. 787-791.
50. Lin, K.-M., et al., Proceedings of the National Academy of Sciences, 2014. 111(2): p. 775-780.
52. Kortmann, J., S.W. Brubaker, and D.M. Monack, The Journal of Immunology, 2015. 195(3): p. 815-819
54. Hu, Z., et al., Science, 2013. 341(6142): p. 172-175.
55. Tenthorey, Jeannette L., et al., Molecular Cell, 2014. 54(1): p. 17-29.
56. Zhang, L., et al., Science, 2015. 350(6259): p. 404-409.
57. Kayagaki, N., et al., Science, 2013. 341(6151): p. 1246-1249.
58. Hagar, J.A., et al., Science, 2013. 341(6151): p. 1250-1253.
60. Broz, P. and V.M. Dixit, Nature Reviews Immunology, 2016. 16: p. 407.
61. Kaparakis-Liaskos, M. and R.L. Ferrero, Nature Reviews Immunology, 2015. 15: p. 375.
62. Meunier, E., et al., Nature, 2014. 509(7500): p. 366-370.
63. Man, S.M., et al., Cell, 2016. 167(2): p. 382-396.e17.
64. Santos, J.C., et al., The EMBO journal, 2018. 37(6): p. e98089.
65. Bian, Z.-M., et al., Investigative Ophthalmology & Visual Science, 2011. 52(12): p. 8646-8656.
66. Rathinam, Vijay A.K., et al., Cell, 2012. 150(3): p. 606-619.
67. Christgen, S., D.E. Place, and T.-D. Kanneganti, Cell Research, 2020.
68. Baker, P.J., et al., European Journal of Immunology, 2015. 45(10): p. 2918-2926