上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
产品资料 – 用于二代测序的 NEBNext® 试剂 – 适用于 Illumina 测序平台/Ultra II DNA & RNA
NEBNext® 酶学转化法 5hmC 甲基化建库试剂盒 收藏
货 号
规 格
价 格(元)
北京库存
上海库存
广州库存
成都库存
苏州库存
武汉库存
#E3350L
96 次反应
37,319.00
无
无
无
无
无
无
#E3350S
24 次反应
9,839.00
无
无
无
无
无
无
Download:
- isoschizomers |
- compatible ends |
- single letter code
相关产品:
NEBNext® 5hmC 甲基化建库酶学转化法模块
#E3365L 96 次反应
#E3365S 24 次反应
NEBNext® 表观遗传建库多样本引物试剂盒 2B(Unique 双端 Index 引物)
#E3392S 24 次反应
NEBNext 表观遗传建库多样本引物试剂盒 3(Unique 双端 Index 引物)
#E3404S 96 次反应
产品特点:
• 卓越的 5hmC 检测灵敏度
• 起始量范围广:0.1 – 200 ng
• 更均一的 GC 覆盖度
• 更高效的建库流程
• E5hmC-seq 和 EM-seq 数据结果可联合分析
• 可单独购买转化模块(NEB #E3365)
概述:
NEBNext® 酶学转化法 5hmC 甲基化建库试剂盒(E5hmC-seq™)是一种在单碱基水平上特异性检测 5hmC 位点的新方法。
通常,使用基于 NEBNext EM-seq 酶学转化或重亚硫酸盐转化生成的 Illumina 文库,可以检测修饰的 5-甲基胞嘧啶(5mC)和 5-羟甲基胞嘧啶(5hmC)。然而,这些方法并不能区分 5mC 和 5hmC。
与 EM-seq 相同,NEBNext® 酶学转化法 5hmC 甲基化建库试剂盒(E5hmC-seq™)使用酶学转化法特异性检测 5hmC 位点,不会像重亚硫酸盐转化法对 DNA 造成损伤,即便 DNA 起始量低至 0.1 ng,也能灵敏地检测 5hmC 位点。
在两步酶法转化过程中,T4-BGT 对 5hmC 进行糖基化,使得 5hmC 在下一步 APOBEC 脱氨基反应中被保护了起来。T4-BGT 不保护 5mC 和未甲基化的胞嘧啶,它们被 APOBEC 脱氨基为尿嘧啶。随后使用 NEBNext® Q5U® 预混液(Q5® 高保真 DNA 聚合酶的改良版)进行扩增,并在 Illumina 平台上进行测序。
用于 EM-seq 和重亚硫酸盐测序的生物信息学分析工具也可用于 E5hmC-seq。可以从 EM-seq 数据中减去 E5hmC-seq 数据,从而确定单个 5mC 和 5hmC 的精确位置。
NEBNext® 酶学转化法 5hmC 甲基化建库试剂盒包含 NEBNext Ultra™ II 文库制备试剂和 E5hmC-seq 接头。请注意试剂盒不包含引物,请单独购买(NEBNext 表观遗传建库多样本引物试剂盒, NEB #E3392, NEB #E3404)
E5hmC-seq 转化原理
产品描述:
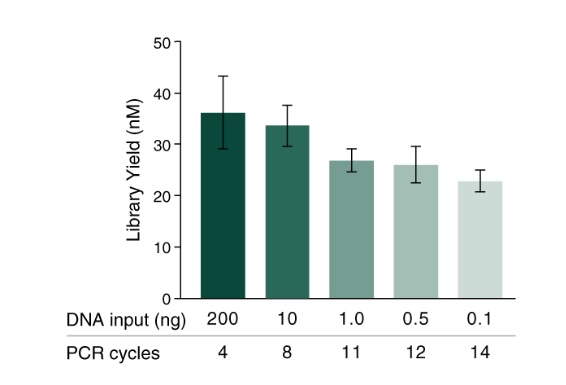
使用不同起始量进行建库,E5hmC 均能获得高产量的文库。
使用 Covaris ME220 仪器将 0.1 ng 至 200 ng 人脑基因组 DNA 打断至 350 bp,并用作 E5hmC-seq 的起始样本,所使用的 PCR 循环数如图所示。使用 Agilent® TapeStation,,High Sensitivity D1000 试剂测定文库产量。显示的数值是四次技术性重复的平均值,误差线表示标准差。结果表明:不同起始量的样本均能获得高产量的 E5hmC-seq 文库。
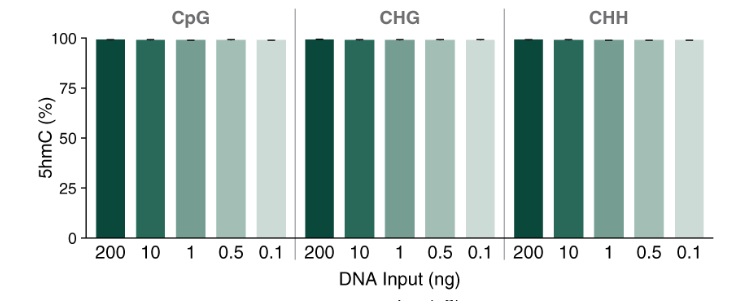
E5hmC-seq 文库 5hmC 检出率高
使用 0.1 ng 至 200 ng 人脑基因组 DNA 制备 E5hmC-seq 文库时,插入所有胞嘧啶完全羟甲基化的 T4 DNA 作为标准品。使用 Covaris ME220 仪器将 DNA 打断至 350 bp,并用作 E5hmC-seq 的起始样本。构建的文库在 Illumina NovaSeq 6000(2 x 150 bases)上进行测序。使用 bwa-meth 将每个文库的 19 亿数据(200 ng、10 ng 和 1 ng)或 7.15 亿 数据(0.5 ng 和 0.1 ng)与人类 T2T、lambda 和 T4 参考基因组的复合基因组进行比对,并使用 MmethylDackel 从中提取甲基化信息。显示的数值是两次技术性重复的平均值,误差线表示标准差。结果表明:T4 DNA 标准品 CpG、CHG 和 CHH 中的 5hmC 检出率 ≥ 98.9%。
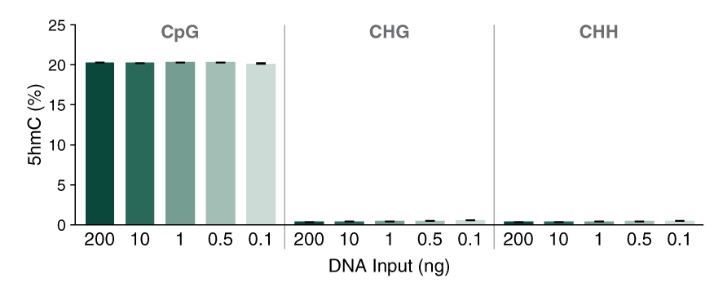
使用不同起始量人脑 gDNA 进行建库,E5hmC 表现出一致的总 5hmC 检测结果。
使用 Covaris ME220 仪器将 0.1 ng 至 200 ng 人脑基因组 DNA 打断至 350 bp,并用作 E5hmC-seq 的起始样本。构建的文库在 Illumina NovaSeq 6000(2 x 150 bases)上进行测序。使用 bwa-meth 将每个文库的 19 亿数据(200 ng、10 ng 和 1 ng)或 7.15 亿 数据(0.5 ng 和 0.1 ng)与人类 T2T、lambda 和 T4 参考基因组的复合基因组进行比对,并使用 MmethylDackel 从中提取甲基化信息。显示的数值是两次技术性重复的平均值,误差线表示标准差。结果表明:不同起始量的样本在 CpG、CHG 和 CHH 中的 5hmC 占比相似。
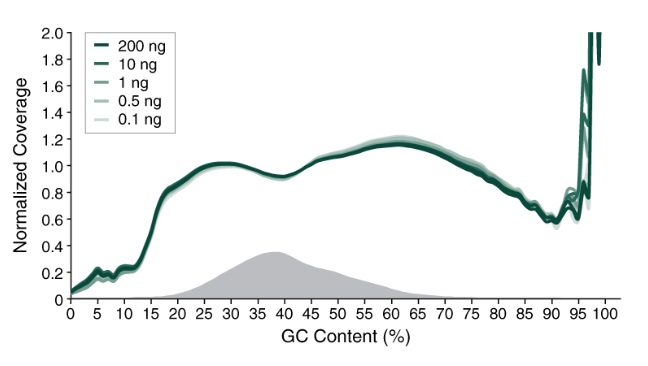
E5hmC-seq 文库 GC 覆盖度均一。
使用 Covaris ME220 仪器将 0.1 ng 至 200 ng 人脑基因组 DNA 打断至 350 bp,并用作 E5hmC-seq 的起始样本。构建的文库在 Illumina NovaSeq 6000(2 x 150 bases)上进行测序。使用 bwa-meth 将每个文库的 19 亿数据(200 ng、10 ng 和 1 ng)或 7.15 亿 数据(0.5 ng 和 0.1 ng)与人类 T2T、lambda 和 T4 参考基因组的复合基因组进行比对。使用 Picard 计算 GC 覆盖度,图中显示不同 GC 含量时(0-100%),标准化后覆盖度的分布情况。将人类 T2T 基因组的 GC 含量分布绘制为直方图。结果表明:不同起始量的样本所制备的 E5hmC-seq 文库皆能获得均一的 GC 覆盖度。
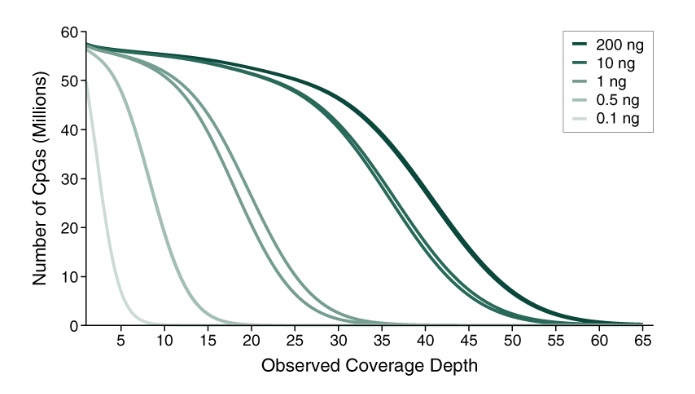
使用不同起始量进行建库,E5hmC-seq 均能获得高 GC 覆盖度。
使用 Covaris ME220 仪器将 0.1 ng 至 200 ng 人脑基因组 DNA 打断至 350 bp,并用作 E5hmC-seq 的起始样本。构建的文库在 Illumina NovaSeq 6000(2 x 150 bases)上进行测序。使用 bwa-meth 将每个文库的 19 亿数据(200 ng、10 ng 和 1 ng)或 7.15 亿 数据(0.5 ng 和 0.1 ng)与人类 T2T、lambda 和 T4 参考基因组的复合基因组进行比对,并使用 MmethylDackel 从中提取甲基化信息,以 methylkit 形式提供报告。使用 CpG 结果,针对所有起始量的 E5hmC-seq 文库生成 CpG 图谱。正义链和反义链上的 CpG 独立计算,在 T2T 基因组中分析出多达 6780 万个可能的 CpG 位点。使用 0.5 ng 至 200 ng 的起始样本时,E5hmC-seq 始终可以覆盖超过 5600 万个 CpG 位点;即使使用 0.1 ng 起始样本时,也可覆盖约 4800 万个位点。
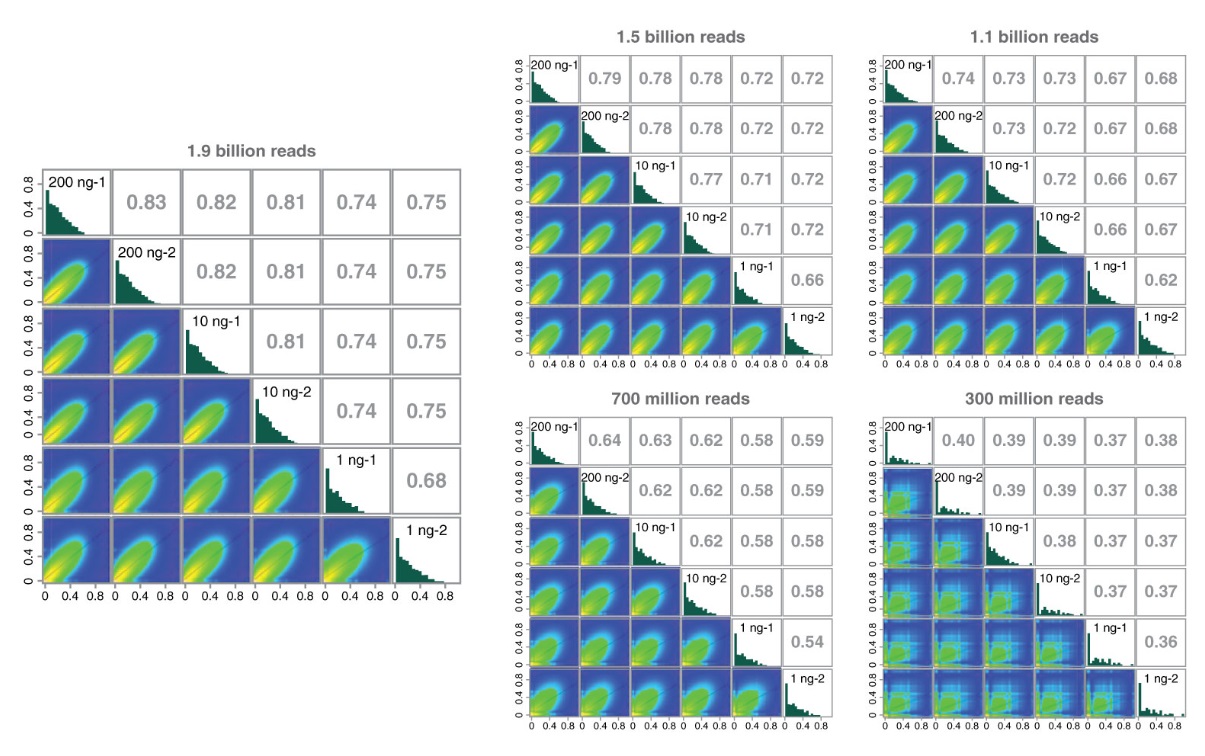
E5hmC-seq 文库在更高测序深度下表现出高相关性。
采用 methyKit 绘制 1 ng、10 ng 和 200 ng 起始量下 E5hmC-seq 文库之间的相关性,19 亿数据测序深度,150-base reads,最小覆盖度为 1X(所有文库均使用 5,650 万个 CpG 位点)。200 ng 和 10 ng 起始量文库的相关性 ≥ 0.81。将 200 ng、10 ng 和 1 ng 的 E5hmC-seq 文库逐步向下采样至约 15 亿、11 亿、7 亿和 3 亿的总 reads,并进行相关性分析。我们观察到,与 11 亿、15 亿和 19 亿 reads 相关性相比,3 亿和 7 亿 reads 的相关性较低。结果表明:由于样本中 5hmC 信号的丰度较低,因此需要对 E5hmC-seq 文库进行深度测序。
